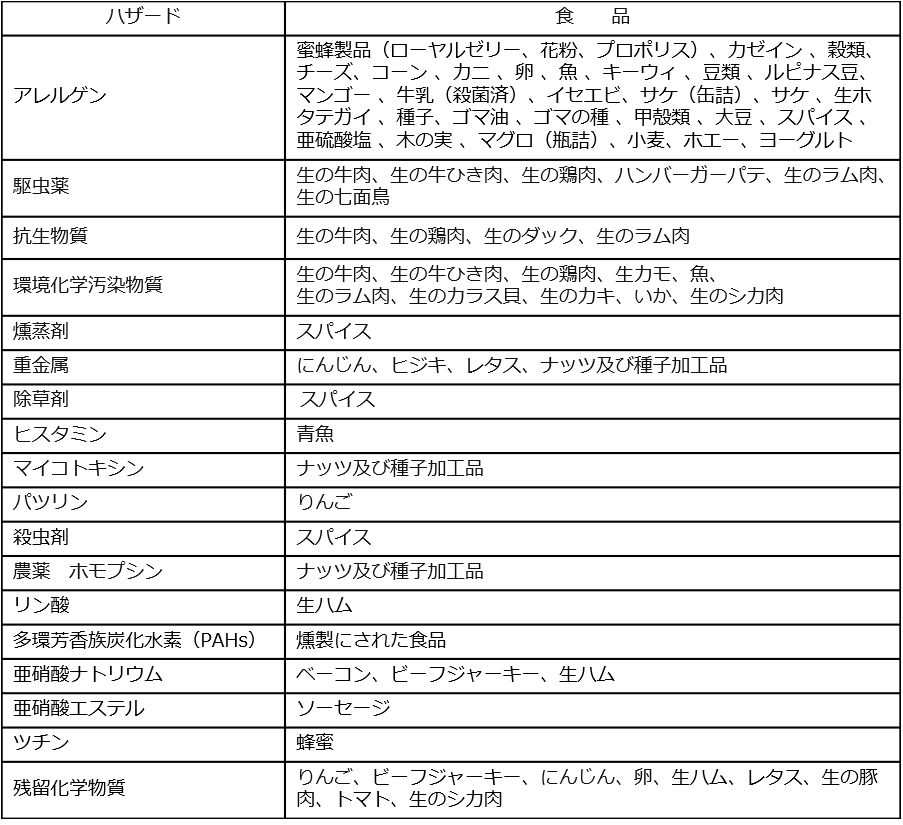
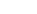予測微生物学
3.新ロジスティックモデルプログラムの使用方法
新ロジスティックモデルプログラムの対象菌種は現在、大腸菌、黄色ブドウ球菌、腸炎ビブリオ、サルモネラおよび一般汚染細菌である。このプログラムの最大の特長は変動温度に対応できる点である。製造から流通、消費までの温度履歴を入力すれば、それに応じた増殖を予測する。
本プログラムを開いたときはVBA機能を使うため「コンテンツの有効化」を有効にする。その使い方の概要はまず、菌種を選択後、初期菌数を入力する。次に温度履歴、つまり温度と時間を1組として温度変化に応じて入力する。実際の温度測定データがあれば、それを使うこともできる。次に予測ボタンを押すと、その温度履歴に対応した予測結果が即座に表れる。牛乳中の黄色ブドウ球菌についてはそのエンテロトキシン生成量も予測される。また、指定した時間での予測した菌数、毒素量も表示できる。以下、実際の使用手順を示す。
3.1. 使用手順
3.1.1 新ロジスティックモデル エクセルマクロプログラム:大腸菌、黄色ブドウ球菌、腸炎ビブリオ版(13)
本プログラムは培地あるいは殺菌した食品(市販牛乳)での測定データから各種温度下での対象微生物の増殖を予測する。
-
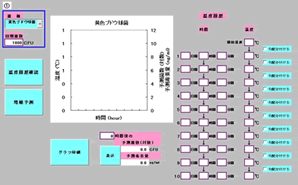
①対象とする菌種をリストボックス中の「大腸菌・黄色ブドウ球菌・腸炎ビブリオ」から選択し、次にその初期菌数を入力する(図1)。この例では、黄色ブドウ球菌(牛乳中)を選択し、その初期菌数を1000 CFU/mlとした。
図1 菌種と初期汚染濃度の入力
-
②温度履歴を入力する(手動)(図2)。最初に開始温度を入力し、次に各温度ステップでの時間と温度を入力する。一定温度のステップではその時間と温度を入力し、経時的な温度勾配のある場合はその温度と経過時間を入れ、「勾配を付ける」というボックスにチェックを入れる。この操作を温度履歴に沿って続ける。ただし、予測適用温度の範囲は15℃から35℃である。
図2 温度履歴の入力(手動)

-
③入力した温度履歴を確認する(図2)。「温度履歴確認」ボタンを押し、入力した温度履歴が正しいかをグラフで確認する。温度履歴曲線は赤線で示される。もし、間違っていれば②に戻って数値を入れ直し、再度「温度履歴確認」ボタンを押して確認する。
-
④「増殖予測」ボタンを押し、温度履歴に対応した増殖予測曲線を得る(図3)。増殖予測曲線は青い線で示される。黄色ブドウ球菌の場合は牛乳中のエンテロトキシンの産生予測量を得ることもできる。予測毒素量は紫色の線で示される。
図3 増殖予測結果

-
⑤指定した時間での予測菌数を知る(図3)。指定した時間を入力し、「表示」ボタンを押すとその時刻における予測菌数が得られる。黄色ブドウ球菌の場合はその時刻におけるエンテロトキシンの予測産生量を知ることもできる。この図では20時間後の値を求めた。
出力した増殖予測曲線を印刷できる。つまり、「グラフ印刷」ボタンを押した後、Excelの通常の操作指示に従って印刷することができる。
デジタル温度記録計などによる連続した温度データも同様に使うことができる(図4)。対象菌種とその初期菌数を入力した後、時間‐温度データを図右側の表に貼り付け、「増殖予測」ボタンを押す。ただし、図4では前述した手動による場合と同じ温度パターンを入力した場合の予測結果を示す。図4 温度履歴の入力(自動)と増殖予測結果

3.1.2 新ロジスティックモデル エクセルマクロプログラム:腸炎ビブリオ版 (14)
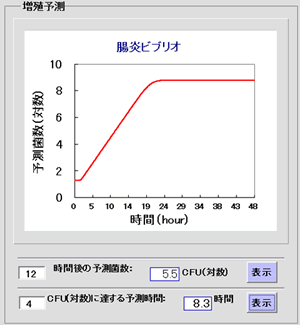
本プログラムは各種の環境条件(温度、塩分濃度、pH)、初期菌数および保存時間における腸炎ビブリオの増殖を予測するプログラムである。本プログラムは培地での測定データから作成されている。使用方法はまず環境条件などを入力し(図5)、次に「増殖予測」ボタンを押すと、その条件下での増殖予測曲線が表示される(図6)。また、指定した時間での予測菌数および指定した菌数に達するまでの予測時間も表示される(図6)。
図5 環境条件(塩分濃度、温度、pH)、保存時間および初期菌数の入力

図6 増殖予測結果:腸炎ビブリオ
3.1.3 新ロジスティックモデル エクセルマクロプログラム:サルモネラ(鶏肉、液卵)(15)
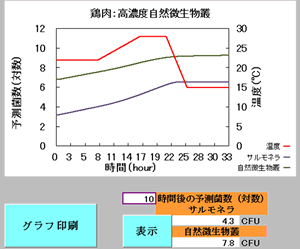
本プログラムは各種温度条件下の鶏ひき肉および液卵におけるサルモネラ(エンテリティディス)と自然汚染微生物叢(一般細菌)の増殖を予測する。鶏ひき肉は一般微生物汚染が高および低レベルの2種類、液卵は殺菌・未殺菌市販液卵の2種類である。鶏ひき肉では上記の予測プログラムと同様に、自然汚染微生物叢レベルを「高」あるいは「低」から選び、次いで菌種をサルモネラ、自然微生物叢あるいはその両方か選ぶ。自然微生物叢のない滅菌鶏肉中でのサルモネラ増殖も予測できる。液卵では、未殺菌液卵と殺菌液卵はそれぞれ自然微生物叢が「高」および「なし」に該当する。また液卵ではサルモネラのみの予測となる。
次いで、温度履歴を入力し、「温度履歴確認」ボタンを押すと、中央画面に温度履歴曲線が表れる(図7)。間違っていれば数値を入れ直し、再度「温度履歴確認」ボタンを押して確認する。
図7 食品の種類、自然微生物叢、予測対象菌種および温度履歴の入力:サルモネラ(鶏肉、液卵)

次いで「増殖予測」ボタンを押すと、入力した条件に応じたサルモネラと自然汚染微生物叢の予測増殖曲線が示される(図8)。
図8 増殖予測結果(サルモネラ(鶏肉、液卵))
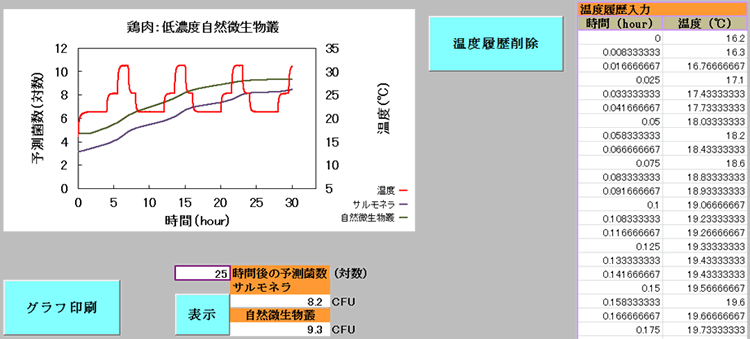
また、前述したエクセルマクロプログラム(大腸菌、黄色ブドウ球菌、腸炎ビブリオ版:温度)と同様、連続測定した温度データを図9(画面右側)のようにコピー・ペーストし、「増殖予測」ボタンを押して増殖予測ができる。
図9 温度履歴の入力(自動)と増殖予測結果(サルモネラ(鶏肉、液卵))
3.1.4 新ロジスティックモデル エクセルマクロプログラム:サルモネラ(牛肉)(11, 12)
本プログラムは各種温度下での牛ひき肉中のサルモネラ(エンテリティディス)と自然汚染微生物叢(一般細菌)の増殖を予測する。このプログラムではサルモネラの初期汚染菌数がある範囲で未知の場合でもその増殖を予測できる(12)。すなわち、このプログラムではサルモネラの初期汚染濃度を2-4 log (cfu/g)の範囲で任意に選ぶことができる。また温度は28℃を超えない範囲で予測できる。
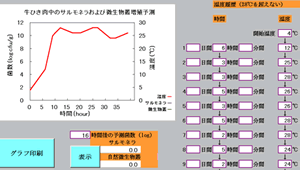
使用方法は新ロジスティックモデル エクセルマクロプログラム(サルモネラ(鶏肉、液卵))と同様である。サルモネラの初期汚染菌数を入力し、予測したい微生物をサルモネラと自然汚染微生物叢から選ぶ。温度を手動入力する場合、「勾配」のボタンは削除し、操作を迅速化してある。温度データを手動入力し、「温度履歴確認」ボタンを押して、温度履歴を確認する。その例を図10に示す。
図10 手動温度入力とその結果:サルモネラ(牛肉)
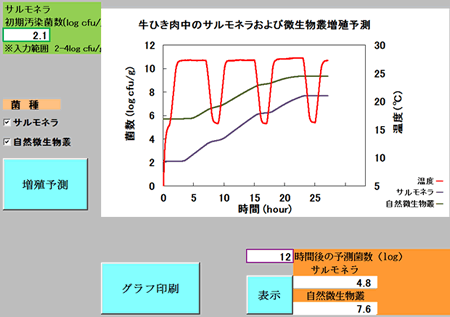
温度履歴を自動入力する場合は上記のプログラム(図9)と同様に、画面右側の2列に時間と温度をコピー・ペーストする。次に、増殖予測ボタンを押すと、図11のような予測結果が得られる。この例では温度履歴を自動入力し、サルモネラ初期汚染菌数を2.1 log(cfu/g)、サルモネラと自然微生物叢の両者の増殖を予測した。さらに、特定の保存時間後の両者の予測菌数も得られる。この例では12時間後の菌数を示す(図11右下)。ただし、牛ひき肉の自然汚染微生物叢(一般細菌)の汚染濃度は一定である。
図11 温度履歴の入力(自動)と増殖予測結果:サルモネラ(牛肉)
資料)
- Whiting, R. C., Buchanan, R. L. (1994): Microbial modeling. Food Technol., 48, 113-120.
- Gibson, A. M., Bratchell, N., Roberts, T. A. (1987): The effect of sodium chloride and temperature on the rate and extent of growth of Clostridium botulinum type A in pasteurized pork slurry. J. Appl. Bacteriol. 62, 479-490
- Baranyi, J., Roberts, T.A. (1994): A dynamic approach to predicting bacterial growth in food. Int.J. Food Microbiol. 23, 277-294.
- Fujikawa, H., Kai, A., Morozumi, S. (2003): A new logistic model for bacterial growth. J. Food Hyg. Soc. Japan. 44, 155-160.
- Fujikawa, H., Kai, A., Morozumi, S. (2004): A new logistic model for Escherichia coli growth at constant and dynamic temperatures. Food Microbiol. 21, 501-509.
- Fujikawa, H., Morozumi, S. (2005): Modeling surface growth of Escherichia coli on agar plates. Appl. Environ. Microbiol. 71, 7920-7926.
- Fujikawa, H., Morozumi, S. (2006): Modeling Staphylococcus aureus growth and enterotoxin production in milk. Food Microbiol. 23, 260-267.
- Zaher,S.M., Fujikawa, H. (2011): Effect of native microflora on the growth kinetics of Salmonella Enteritidis strain 04-137 in raw ground chicken. J. Food Prot.74,735-742.
- Sakha, M.Z., Fujikawa, H. (2012): Growth kinetics of Salmonella Enteritidis in pasteurized and unpasteurized liquid egg products. Biocont. Sci. 17, 83-90.
- Sakha, M.Z., Fujikawa, H. (2013): Prediction of Salmonella Enteritidis in pasteurized and unpasteurized liquid egg products with a growth model. Biocont. Sci. 18, 89-93.
- Sabike,I.I., Fujikawa, H.,Edris, A.M. (2015): The growth kinetics of Salmonella Enteritidis in raw ground beef. Biocont. Sci.20, 185-192.
- Fujikawa, H., Sabike,I.I., Edris, A.M. (2015): Prediction of the growth of Salmonella Enteritidis in raw ground beef at various combinations of the initial concentration of the pathogen and temperature. Biocont. Sci.20, 215-220.
- 藤川浩他 2006 各種温度下における微生物増殖予測プログラムの開発 食衛誌 47, 288-292.
- Fujikawa, H., Kimura, B., Fujii, T. (2009): Development of a predictive program for Vibrio parahaemolyticus under various environmental conditions. Biocont. Sci. 14, 127-131.
- Fujikawa, H., Sakha, M.Z. (2013): Development of a predictive program for Salmonella Enteritidis growth in ground chicken and liquid egg products. Biocont. Sci. 18, 175-179.